
やること:
仮想実験室
分子生物学の実験を仮想的に再現し、研究の効率を高めることを目的としています。
主に次のことができます。
- プライマーの設計
- クローニングのために制限酵素サイトやGatewayのattB1配列をフレームを合わせて設計することは骨の折れることです。 仮想実験室ではアミノ酸配列を見ながら簡単な操作でプライマーを設計できます。
- DNAの特定の塩基を調べるためにプライマーを設計する必要があるときや、特定の塩基配列が存在するかどうかを調べるためにプライマーを設計することもよくあります。 このようなときプライマーが結合する位置はそれほど厳密にする必要はありませんが、PCR産物のおおよその大きさとプライマーの相対的な位置を工夫する必要があります。 仮想実験室では「ここを増やしたい」プライマーをお手軽に作成できます(予定)。
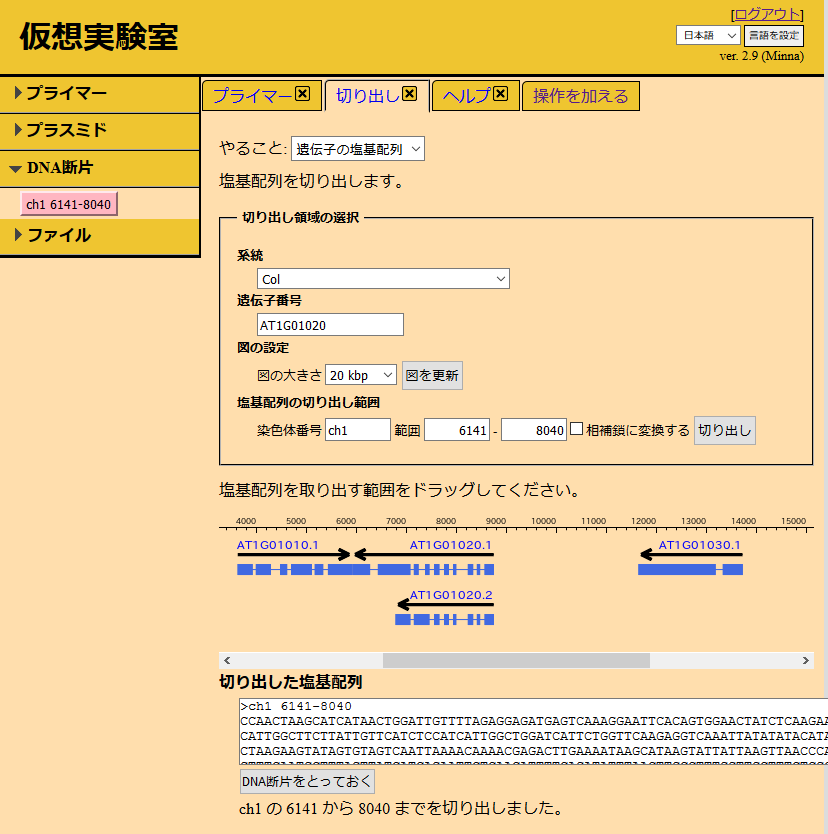
- 塩基配列の取り出し
- Mitsucalのデータベースから塩基配列を自在に切り出します。 染色体の塩基配列から目的の部分を取り出すのは面倒な作業です。 仮想実験室では遺伝子地図を見ながら簡単な操作で塩基配列を取り出します。
- コンストラクトづくりの手伝い
- 設計したプライマーを使ってPCRをしたときに得られるDNA断片を取得します。
- それをベクターにクローニングします。
- できたプラスミドを制限酵素で切って確認します。
- ちゃんとタンパク質になるかどうかチェックします。(予定)
- シークエンスデータ(.ab1ファイル)と比較して、ミスがないかどうか確認できます。(実装中)
仮想実験室の使い方
仮想実験室のページは下のようになっていて、データを取っておく左の部分(冷蔵庫)と、様々な処理を行う右の部分(試験管)からなっています。
冷蔵庫
冷蔵庫には「プライマー」、「プラスミド」、「DNA断片」をいれておく箱があり、箱の名前をクリックすると中身を表示します。
冷蔵庫にある材料は試験管のフォームにドロップすることで反応に投入することができます。
冷蔵庫にある材料をクリックすると、その名前を変更したり、捨てたりすることができます。
冷蔵庫の一番下のファイルには様々なツールがあります。作成したデータをまとめてファイルとして保存したり、保存したファイルをアップロードして仮想実験を再現したりすることができます。(予定)
試験管
試験管は上部にタブがあり、クリックで切り替えることができます。
「操作を加える」をクリックすると試験管を増やすことができます。
試験管でできたものは「プラスミドをとっておく」などのボタンをクリックすると冷蔵庫に入れることができます。遺伝子クローニングの手順
シロイヌナズナのPATL2遺伝子(AT1G22530)をクローニングしてGFP融合タンパク質を植物で発現させる実験を例に仮想実験室の使い方を説明します。
1. プライマー設計領域の塩基配列を取り出す
- 仮想実験室に新しい試験管を加えます。やることの一覧から「遺伝子の塩基配列」を選択します。
- 「切り出し領域の選択」フォームの「系統」からArabidopsis thalianaのColを選択します。 「遺伝子番号」にAT1G22530を入力し、「図を更新」をクリックします。 遺伝子が大きくて範囲が選択しにくいときは図の大きさを適当に変更し、図を更新してください。
- クローニングしたい範囲をマウスでドラッグしてください。 ドラッグすると染色体番号と範囲が入力されます。 PATL2は相補鎖にコードされているので、「相補鎖に変換する」にチェックを入れます。 「切り出し」ボタンをクリックしてください。
- 切り出した塩基配列が表示されます。 「DNA断片をとっておく」ボタンをクリックします。
- 冷蔵庫のDNA断片箱にボタンが増えます。これをクリックし、出てきたメニューの編集をクリックします。 名前をPATL2 genomeとして更新ボタンを押します。
2. クローニングのためのプライマー設計
- 仮想実験室に新しい試験管を加えます。やることの一覧から「塩基置換導入」を選択します。
- 塩基配列を入力のフォームに上記で作成したPATL2 genomeをドラックします。 塩基配列が入力され、設計テーブルが更新されます。
- 設計テーブルの2行目に入力した塩基配列の一部が表示されています。 同じ行の右にあるテキストボックスに数字を入力するか、スピナー(上下の三角形のボタン)をクリックしてプライマーを設計する領域を表示させます。 スピナーの右のテキストボックスに塩基配列を入力すると、その配列がある場所まで移動します。
- 設計テーブルの3行目はプライマーを設計する領域を示します。位置と長さを調節してください。 プライマーの長さ、Tmが下に表示されます。
- GatewayのBP反応でクローニングするために、attB1配列を付加します。 4行目の置換後の配列の右の選択ボックスからattB1 (25bp)を選択します。 プライマーの設計位置と置換後の配列の位置を調節します。 プライマーの設計領域は置換後の配列を含めるようにしてください。 そのとき、Tmはプライマー全体のTmを計算していることに注意してください。
- 適当なプライマーが設計できたら、「プライマーをとっておく」ボタンをクリックし、冷蔵庫に保管します。 名前を attB1-PATL2 とします。
-
同様に下流側(アンチセンス)のプライマーを設計します。
注意点は以下の通りです。
- PATL2のC末端のアミノ酸配列はLYRSKTQAです。 これを使って設計テーブルに表示される塩基配列を調節してください。
- attB2をプライマー設計領域の右側に置いてください。
- アンチセンス側を設計にチェックをいれてください。
- アンチセンスのプライマーはフレームを合わせる必要があるので、attB2によって置換された塩基配列がPAFLYKVと翻訳されるように位置を決めてください。
- 「プライマーをとっておく」ボタンをクリックし、冷蔵庫に保管します。 名前を attB2-PATL2 とします。
3. PCR
- 仮想実験室に新しい試験管を加えます。やることの一覧から「PCR」を選択します。
- 反応条件フォームのプライマー1に先ほど作成したattB1-PATL2をドロップします。 同じくプライマー2にattB2-PATL2をドロップします。
- 系統はArabidopsis thalianaのColを選択し、種類は染色体DNAを選択します。 鋳型DNAは空にしておいてください。 「PCR」ボタンをクリックします。
- ch1の一部にattB1とattB2の配列が結合した塩基配列が表示されます。 「PCR産物をとっておく」ボタンをクリックし、冷蔵庫に保管します。 名前を attB-PATL2とします。
4. BP反応
- 仮想実験室に新しい試験管を加えます。やることの一覧から「BP反応」を選択します。
- 反応条件のベクター選択ボックスでpDONR201を選択します。 ベクターの塩基配列は空にしておいてください。
- 先ほど作成した attB-PATL2をインサートにドロップします。 PCR産物の塩基配列が表示されます。
- 「BP cloning」ボタンをクリックします。
- 生成物に BP_product が表示されます。 P1からB1への組換えとB2からP2への組み換えが起きていれば成功です。 「プラスミドをとっておく」ボタンをクリックし、冷蔵庫に保管します。 名前を pDONR-PATL2とします。
5. シークエンス (未完成)
- 仮想実験室に新しい試験管を加えます。やることの一覧から「シークエンス」を選択します。
- 解析の参照配列に pDONR-PATL2 をドロップします。
- pDONR-PATL2をシークエンスした ab1 ファイル(波形データ)をファイル送信にドロップします。
- pDONR-PATL2の配列と実際のデータに違いがないかどうかを調べます。
6. LR反応
- 仮想実験室に新しい試験管を加えます。やることの一覧から「LR反応」を選択します。
- 反応条件のベクターとしてpGWB404を選択します(プロモーター無し、C末端sGFP付加)。
- エントリークローンにpDONR-PATL2をドロップします。
- 「LR cloning」をクリックします。
- 生成物に LR_product が表示されます。 R1からL1への組換えとR2からL2への組換えが起きていれば成功です。 「プラスミドをとっておく」ボタンをクリックし、冷蔵庫に保管します。 名前を pGWB404-PATL2とします。
7. 制限酵素の切断パターンを調べる (オプション)
- 仮想実験室に新しい試験管を加えます。やることの一覧から「制限酵素処理」を選択します。
- 反応条件の基質に pGWB404-PATL2 をドロップします。 基質の形状の「環状」を選択します。 制限酵素欄にHincIIと入力します。 「切断」ボタンをクリックします。
- 制限酵素サイトの情報、断片の長さを確認します。 実際の結果と一致しているかどうかを検討してください。